Så kommer då bloggens utlovade inlägg ni läsare valde. Det har tagit tid att samla in, sortera och välja ut de fakta som behövs för att förstå innebörden av det ni kommer få läsa sist i detta inlägg (universitetets forskning). Kärnan i inlägget kommer vara QPI (Quantitative Phase Imaging) som är den teknik PHI konstruerat sitt instrument HoloMonitor på. För att förstå vad som differentierar QPI mot annan mikroskopiteknik som ljus,elektron,atomic force-mikroskopi börjar jag med att konstatera att celler är rackarns svåra att observera då de mer eller mindre är osynliga för det mänskliga ögat (även sett i ett trad ljusmikroskop). Därför har forskare tvingats färga in cellerna för att kunna urskilja dessa från varandra och bakgrunden. Det har nackdelar då cellerna iochmed infärgningen startar sin dödsprocess samtidigt som infärgningen påverkar cellernas normala utveckling som ex rörlighet och förändring i form och storlek.
I hur stor utsträckning infärgningen påverkar resultat av cellobservationerna är väl egentligen inte vederlagt då forskarna ofta tar en ny batch av samma celltyp när den första har gjort sitt (cellerna har påverkats för mkt av infärgningen och är då inte användbara längre).Fungerar när man har cellodling och därmed obegränsad tillgång. Av lättförstådda skäl fungerar det inte när man enbart har tillgång till begränsat antal celler. Den kloke läsaren tänker nu direkt på den Regenerativa forskningen,och det görs med all rätta.
QPI som teknik fungerar så att en ljusstråle (laser) belyser cellerna under en kort tid. När det sker fångar tekniken upp den lilla fördröjning/förändring av ljusstrålen som sker när strålen passerar genom cellen. Fördröjningen kallas på fackspråk för fasförskjutning,phase på engelska Sen tar en datoralgoritm över och omvandlar fördröjningen/förändringarna till celltjocklek i ett första skede. I nästa moment har programvaran skapat en topografisk bild utifrån första momentet. Slutligen färglägger systemet (datorn) resultaten och skapar ex en enskild bild på cellen eller en utzoomning där man ser hela cellpopulationen. Samtidigt som omvandlingen skett sprattlar cellerna vidare i kärlet lyckligt ovetande att en dator nu gjort dem synliga. Datorn följer då cellernas utveckling med att filma dem och se vad de har för sig (utan att bli infärgade eller på annat sätt manipulerade).
Här en bild på proceduren
Se exempelvis kinesernas flitiga användande av HoloMonitor för den svårbotade cancern glioblastoma.
Läs mer om tekniken på PHI`s hemsida : Quantitative Phase Imaging
För att ni ska förstå att tekniken hunnit ifatt verkligheten (eller var det tvärtom?..hmm) går jag till Nature och låter dem berätta om utvecklingen. 2018 skrev de följande (översättning) :
Kvantitativ fasavbildning (QPI) har visat sig vara en värdefull metod för att undersöka celler och vävnader. QPI fungerar på omärkta prover och är som sådan ett komplement till etablerad fluorescensmikroskopi, uppvisar lägre fototoxicitet och ingen fotoblekning. Eftersom bilderna representerar kvantitativa kartor över optiska väglängdsfördröjningar som introduceras av provet, ger QPI ett objektivt mått på morfologi och dynamik, fritt från variabilitet på grund av kontrastmedel. På grund av de enorma framsteg som bevittnats, särskilt under de senaste 10–15 åren, har ett antal tekniker blivit tillräckligt tillförlitliga och överförts till biomedicinska laboratorier. Kommersialiseringsarbetet pågår och som ett resultat av detta övergår QPI-området nu från ett teknikutvecklingsdrivet till ett applikationsfokuserat område.
2023 har vi kommit lite längre och Nature skriver följande (översättning) :
Kvantitativ fasavbildning (QPI) har utvecklats för att avgränsa strukturella och dynamiska egenskaper hos transparenta celler och tunna vävnader genom att utforska den inneboende bildkontrasten från brytningsindex (RI) och tjockleksvariationer. Som en märkningsfri avbildningsmetod har QPI möjliggjort många unika biomedicinska studier, såsom att belysa celltillväxtmekanismer genom att kvantifiera massförändringar på femtogramnivå, särskiljande tillstånd av blodsjukdomar och sondera elektrisk aktivitet genom att mäta cellmembranfluktuationer i nanometerskala. Dessutom har en distinkt RI-kontrast mellan normala och onormala celler och vävnader rapporterats för olika sjukdomar, vilket visar potentialen i att använda RI som en inneboende diagnostisk biomarkör. Optisk diffraktionstomografi (ODT) är en förlängning av QPI som möjliggör volymetrisk avbildning av biologiska prover genom att kartlägga deras tredimensionella (3D) RI-kartor, vilket ytterligare främjar studier inom cellorganelldynamik farmakologi immunologi neurovetenskap och patologi vid infektionssjukdoma. I ODT mäts först multipla kvantitativa fasbilder under olika förhållanden, inklusive belysningsvinkel eller våglängdsskanning, transponera sample lateralt med en linjefokusstråle eller axiellt med koherensreglerande effekt. Genom att lösa ett inversspridningsproblem med hänsyn till optisk diffraktion erhålls RI-rekonstruktioner från de komplexa fältmätningarna, vilket gör det möjligt att observera högupplösta egenskaper i levande celler.
Förmågan att avbilda tjocka biologiska vävnader in vivo är avgörande för många banbrytande biologiska studier och kliniska diagnostiska tillämpningar.
För 3 veckor sen kom den första? studien om hur praktiskt användbar AI (Artificiell Intelligens) i konkreta situationer kommer vara tillsammans med QPI. Nature skriver följande (översättning) :
Kvantitativ fasavbildning (QPI), integrerad med artificiell intelligens, möjliggör snabb och märkningsfri undersökning av fysiologi och patologi hos biologiska system. Denna översikt presenterar principerna för olika tvådimensionella och tredimensionella etikettfria fasavbildningstekniker som utnyttjar brytningsindex som en inneboende optisk bildkontrast. I synnerhet diskuterar vi artificiella intelligensbaserade analysmetoder för biomedicinska studier, inklusive bildförbättring, segmentering av cellulära eller subcellulära strukturer, klassificering av typer av biologiska prover och bildöversättning för att tillhandahålla subcellulär och histokemisk information från etikettfria fasbilder. Vi diskuterar också fördelarna och utmaningarna med kvantitativa fasavbildningsanalyser som möjliggörs av artificiell intelligens, sammanfattar de senaste anmärkningsvärda tillämpningarna inom livsvetenskaperna och täcker potentialen för detta område för grund- och industriell forskning inom livsvetenskaperna.
Koreanska forskare har tidigare publicerat en studie om QPI + AI. Man kan inte ta miste på deras entusiasm över vilka möjligheter som öppnas. Länk
— Recent advances in quantitative phase imaging (QPI) and artificial intelligence (AI) have opened up the possibility of an exciting frontier. The fast and label-free nature of QPI enables the rapid generation of large-scale and uniform-quality imaging data in two, three, and four dimensions. Subsequently, the AI-assisted interrogation of QPI data using data-driven machine learning techniques results in a variety of biomedical applications. Also, machine learning enhances QPI itself. Herein, we review the synergy between QPI and machine learning with a particular focus on deep learning. Further, we provide practical guidelines and perspectives for further development.
Amerikanska NIH publicerade för 3 veckor sen en studie där forskare fått fram än mer info från QPI i sina studier. Abstraktet (översättning) :
Kvantitativ fasavbildning (QPI) har visat sig vara ett oumbärligt verktyg inom biomedicin, som erbjuder möjligheten att få kvantitativa kartor över fasförändringar på grund av optiska väglängdsfördröjningar utan behov av kontrastmedel. Dessa kartor ger värdefull information om cellulär morfologi och dynamik, ostörd av införandet av exogena ämnen. I denna översikt presenteras en sammanfattning av nyligen genomförda studier som har fokuserat på att belysa tillväxtdynamiken hos enskilda celler med hjälp av QPI. Specifikt undersöks undersökningar av cellulära förändringar som uppstår under mitos, differentiering av cellulära organeller, bedömning av distinkta celldödsprocesser (dvs. apoptos, nekros och onkos) och den exakta mätningen av levande celltemperatur. Dessutom lyfts de fängslande tillämpningarna av QPI inom tragnostik, där dess potential för transformativ inverkan visas upp på ett framträdande sätt. Slutligen beskrivs de utmaningar som måste övervinnas för att den ska kunna antas i större utsträckning och integreras framgångsrikt i biomedicinsk forskning.
Braighta forskare har grottat ner sig i tekniken och funnit att QPI är användbar inte bara för cellstudier utan även för enzymforskning.Nu snackar vi ner på molekylnivå. Amerikanska NIH skriver följande (översättning) :
Kvantitativ fasavbildning (QPI) är en kraftfull optisk avbildningsmodalitet för etikettfri, snabb och tredimensionell (3D) övervakning av celler och vävnader. Molekylär avbildning av viktiga intracellulära biomolekyler såsom enzymer är dock fortfarande ett i stort sett outforskat område för QPI. Här introducerar vi ett fundamentalt nytt tillvägagångssätt genom att designa QPI-kontrastmedel som möjliggör känslig detektion av intracellulära biomolekyler. Vi rapporterar en ny klass av bio-ortogonala QPI-nanoprober för in situ högkontrastrefraktivt index (RI) avbildning av enzymaktivitet. Nanosonderna har kiseldioxidnanopartiklar (SiO2 NPs) med högre RI än endogena cellulära komponenter och ytförankrade cyanobenzotiazolcystein (CBT-Cys) konjugerade enzymresponsiva peptidsekvenser. Nanosonderna är specifikt aggregerade i celler med målenzymaktivitet, vilket ökar intracellulär RI och möjliggör exakt visualisering av intracellulär enzymaktivitet. Vi föreställer oss att denna generella design av QPI-nanoprober kan öppna dörrar för rumslig-temporal kartläggning av enzymaktivitet med direkta implikationer för sjukdomsdiagnos och utvärdering av den terapeutiska effekten.
---------------------------------------------------------------------------------------------------------
När kommer den infon undrar kanske den ivrige? Innan ni får läsa om deras gärning (cancerforskning) levererar bloggen en rykande het (och aktuell) amerikansk forskning inom ett helt annat område.
Kardiostudier (hjärta) att sortera under den Regenerativa medicinen. Forskning utförd med tadaaa..HoloMonitor...och då naturligtvis QPI.
För att det här inlägget inte ska bli alldeles för långt går jag inte in på forskningens innehåll utan klipper enbart in forskarnas användande och uttalanden om HoloMonitor.
ABSTRACT
Cardiac regeneration in newborn rodents depends on the ability of pre-existing cardiomyocytes to proliferate and divide. This capacity is lost within the first week of postnatal development when these cells rapidly switch from hyperplasia to hypertrophy, withdraw from the cell cycle, become binucleated, and increase in size. How these dynamic changes in size and ploidy impact cardiomyocyte proliferative potential is not well understood. In this study, we innovate the application of a commercially available digital holographic imaging microscope, the Holomonitor M4, to evaluate the proliferative responses of mononucleated diploid and binucleated tetraploid cardiomyocytes. This instrument coupled with the powerful Holomonitor App Suite software enables long-term label-free quantitative three-dimensional tracking of primary cardiomyocyte dynamics in real-time with single-cell resolution. Our digital holographic imaging results provide direct evidence that mononucleated cardiomyocytes retain significant proliferative potential as most can successfully divide with high frequency.
Materials and Methods (urval)
Digital holographic time-lapse imaging
The Holomonitor M4 digital holographic microscope was purchased from Phase Holographic Imaging (PHI, Sweden) and placed directly within a Heracell 150 CO2 incubator (Thermo Scientific). For digital holographic imaging experiments, rat cardiac cells were cultured and treated with chemicals in 6-well plates (83.3920, Sarstedt) covered with HoloLids (PHI, Sweden) to stabilize imaging conditions. Live rat cardiac cultures were imaged on the Holomonitor M4 for 48 to 72 hours with time points taken every 20 min at 37°C and 5% CO2. Prior to imaging on the Holomonitor M4, the plate was covered with HoloLids and images were acquired every hour for 5 days at 37°C and 5% CO2.
Quantitative cell size and motility analyses
Time-lapse recordings were analyzed in the Holomonitor App Suite software (PHI, Sweden) using the “In-depth Analysis: Single Cell Tracking” feature to segment, trace, and quantify single cell dynamics. The following segmentation thresholds were applied to generate the results shown in Figures 2, 3, and Supplementary Figure 1: auto-minimum error method, adjustment 128, minimum object size 20. The following segmentation thresholds were applied to generate the results shown in Figures 4, 5, and Supplementary Figure 2: auto-minimum error method, adjustment 131, minimum object size 18. Cardiomyocytes were qualitatively distinguished from non-cardiomyocytes based on their larger size and limited motility as shown in Figure 2. The Holomonitor App Suite software was then used to track individual cells and optical volume, thickness, and motility measurements were taken at the specified time points. Care was taken to ensure that all cells analyzed were well-isolated and the accuracy of cell segmentation was manually verified on each imaging frame.
 |
| Three-dimensional digital holographic imaging of cultured primary cardiac cells with the Holomonitor M4 |
(A) Primary cardiac cultures are placed onto the Holomonitor M4 housed within a standard CO2 incubator. A diode laser is divided into a reference beam and an object beam that passes through the sample, which creates a phase shift. An interference pattern hologram is generated by merging of the object and reference beams. This is recorded by an image sensor to reconstruct a phase image. (B) Example images of an interference pattern hologram and three-dimensional reconstructions of cultured primary rat cardiomyocytes as visualized on the Holomonitor M4. Created with BioRender.com.
The HoloMonitor M4 enables direct quantitative three-dimensional label-free imaging of any adherent cells in culture. Cardiomyocytes make up most of the heart’s volume due to their larger size but are outnumbered by smaller cardiac fibroblasts and endothelial cells (Baum & Duffy, 2011). We therefore evaluated if our label-free digital holographic imaging approach could differentiate primary cardiomyocytes from non-cardiomyocytes in mixed primary cardiac cultures based on their unique three-dimensional morphologies and dynamics.
To define which primary cardiac cells observed on the Holomonitor M4 corresponded to cardiomyocytes, we first live-imaged the culture using digital holographic microscopy, fixed the cells, immunostained against the cardiomyocyte-specific marker α-actinin, and then re-imaged the same field by conventional fluorescence microscopy (Fig. 2A-B). The thicker and larger cells identified on the Holomonitor M4 most strongly correlated with α-actinin-positive cardiomyocytes (Fig. 2B-C).
These data demonstrate that our label-free digital holographic imaging approach can reliably distinguish primary neonatal rat cardiomyocytes from non-cardiomyocytes based on their larger size and limited motility. We additionally found that these criteria can be similarly applied to identify primary mouse cardiomyocytes as well (Fig. S1), broadening the applicability of this approach.
 |
| Label-free digital holographic microscopy identifies primary rat cardiomyocytes based on their larger size and limited motility |
(A-B) The same imaging field of fixed postnatal day 2 (P2) rat cardiac cells visualized on a phase contrast fluorescence microscope (A) or live on the Holomonitor M4 (B). Cardiomyocytes (CM) are α-actinin-positive (A, white arrowheads) and larger (B, white arrowheads) while non-CMs are α-actinin-negative (A, green arrowheads) and smaller (B, green arrowheads). DAPI labels all nuclei. White asterisks highlight an α-actinin-negative non-CM that appears thicker. (C-E) Representative three-dimensional reconstructions of a CM and non-CM (C) and quantifications of their maximum optical thicknesses (D) and optical volumes (E). (F-H) Motility analysis of CMs and non-CMs. (F) Representative segmentation and tracking of a CM (red outline) and non-CM (blue outline) over 12 hours. Cell trajectories are shown as a single solid line for each cell. (G) Overlayed trajectories of CMs (n=7) and non-CMs (n=7) over 12 hours. All cell traces begin at the (0,0) coordinate. (H) Total distance traveled by each cell over 12 hours. 150 CMs and 151 non-CMs from postnatal day 2 (P2) hearts were analyzed in D, E, and H. Values are reported as mean ± standard deviation. Student’s t-test; ****, P < 0.0001.
A powerful feature of the Holomonitor App Suite software is its image segmentation capabilities that enable three-dimensional tracking and analysis of single-cell dynamics (Fig. 2F).
DISKUSSION (översättning)
I takt med att fördelarna med digital holografisk avbildning gradvis erkänns, har flera tillämpningar av Holomonitor M4 nyligen rapporterats i olika sammanhang såsom sårläkning, cellrörlighet, tredimensionell odling av celler i matris och optogenetik. Tillämpningen av digital holografisk avbildning inom kardiovaskulär forskning har dock hittills varit begränsad. Mångsidigheten hos detta instrument kan ha stor potential att öka vår cellulära förståelse för kardiomyocythypertrofi, migration, svar på ischemi-reperfusion och intercellulära interaktioner. I denna studie uppfinner vi först en ny tillämpning av denna teknik för att utvärdera dynamiska förändringar i kardiomyocytstorlek under hypertrofisk tillväxt och som svar på en proliferativ stimulans.
Nuvarande metoder för att direkt utvärdera kardiomyocytstorlek har var och en unika experimentella begränsningar. Till exempel misslyckas tvådimensionella mätningar av kardiomyocytytan med att ta hänsyn till förändringar i celltjocklek och mer direkt utvärdera cellspridning. Billräknare och flödescytometri ger populationsbaserade mätningar men löser inte encellsdynamiken. Och tredimensionell konfokal avbildning kräver vanligtvis cellfixering och optisk snittning med kraftfulla lasrar, vilket begränsar långsiktig avbildning av levande kardiomyocyter.
Samspelet mellan kardiomyocytstorlek, ploidi, och cellcykelreglering under denna utvecklingsövergång är inte väl förstått. Därför är vår unika tillämpning av digital holografisk avbildningsteknik relevant för att förstå de cellulära mekanismer genom vilka däggdjurshjärtan förlorar sin förmåga till hjärtregenerering kort efter födseln.
-----------------------------------------------------------------------------------------------------------------
Ok,har ni nu blivit övertygade om QPI och HoloMonitor som ett framtida instrument att användas av många av världens forskare? Om inte till 100% kommer då slutligen infon om det amerikanska universitetet och vad de är på gång med. PHI`s partner Huntsman Cancer Institute...
Quantifying epithelial-mesenchymal transition
PHI is working with scientists at the Huntsman Cancer Institute (HCI) in the battle against cancer. Supported by PHI, Dr. Robert Judson-Torres and his team at HCI aim to develop methods to quantify the phenomena known as epithelial-mesenchymal transition.
HCI is a National Cancer Institute-designated Comprehensive Cancer Center, supported by the National Institutes of Health. "
....samarbetar med Zangle Lab (Utah Cancer Dynamics Laboratory),en avdelning hos UTAH University där även Huntsman Cancer Institute huserar.
Forskare på Zangle Lab bedriver studier om personligt anpassade läkemedel för cancerpatienter. Vi vet sen tidigare att HoloMonitor används för att testa matchning mellan en specifik cancer och idag aktuella läkemedel. Cancerceller studeras tillsammans med substrat från läkemedlet och resulterar förhoppningsvis i ett brett fungerande cancerläkemedel för den kategorin cancerpatienter.
Alltså individuell matchning som även kallas "funktionell precisionsmedicin".
Det finns idag metoder att skräddarsy medicin till patient 1.Men då är tiden för att få fram rätt läkemedel till patient 1 mellan 1-8 månader. Det med dagens metoder alltså. 1-8 månader hjälper ytterst få cancerpatienter då eländet har en förmåga att växa och snabbt utvecklas till.....det är för sent.
Zangleforskarna har då kikat på QPI som helt ny metodik att se hur patient 1 cancerceller reagerar mot cancerläkemedlet X. QPI är i detta fall med användande av HoloMonitor och medföljande App Suite. Det antagandet gör jag då instrumentet finns och används på universitetet (Se Userlistan) samt PHI`s nära samarbete med Huntsman. Jag låter forskarna själva berätta om sin forskning (översättning) :
Vi undersöker användningen av mätningar av cellmassa och massfördelning via kvantitativ fasavbildning (QPI) för att utveckla nya metoder för att studera och behandla mänskliga sjukdomar, inklusive cancer. Vårt arbete inkluderar samarbeten med forskare vid University of Utah, Huntsman Cancer Institute och andra institutioner. Kod från dessa och andra projekt finns på GitHub.
Funktionell precisionsmedicin
Nuvarande metoder för att mäta tumörcellers svar på kemoterapi kräver dock 1-8 månader för att få resultat, vilket gör dessa opraktiska för patientvård.
Vi kan sedan poängsätta potentiella terapier baserat på hur mycket de bromsar eller stoppar denna tillväxt. Detta gör det möjligt för QPI att snabbt mäta enskilda cellers svar på terapi, utan att kräva märkning, och med ett relativt litet antal celler, vilket minskar behovet av kostsam och tidskrävande cellexpansion, vilket minskar den totala analystiden och kostnaden.
Länk till sidan med engelsk originaltext.
Detta inlägg är mastigt att beta sig igenom,men jag rekommenderar alla läsare att ta sig en kopp kaffe och sakta läsa hela texten så ni förstår sammanhanget, hur QPI har möjlighet att förändra forskning och även medverka till att ta fram individuella läkemedel för individuella cancerpatienter. Sistnämnda kommer definitivt förändra cancervård i grunden om Zangleforskarna når ända fram.
Uppgift : fundera på hur många sjukhus,kliniker med cancervård det finns i världen som kan tänkas vara intresserade av att snabbt kunna sätta in mest effektivt läkemedel för sina patienter. Det som krävs för dessa är då att införskaffa ett lämpligt instrument som ger dem svaret. Sannolikheten för att det instrumentet kan vara HoloMonitor torde vara hyfsat stor då metodiken bygger på det instrument Zangleforskarna själva använt för att göra denna upptäckt.
Summasummarum: Efter genomläsning (gärna fler ggr) hoppas jag att ni läsare ser potentialen i QPI och det instrument som levererar de resultat tekniken möjliggör : HoloMonitor.
Mvh the99
Ps. Man får gärna sprida detta inlägg till andra fora eller läsare som kan vara intresserade av PHI.
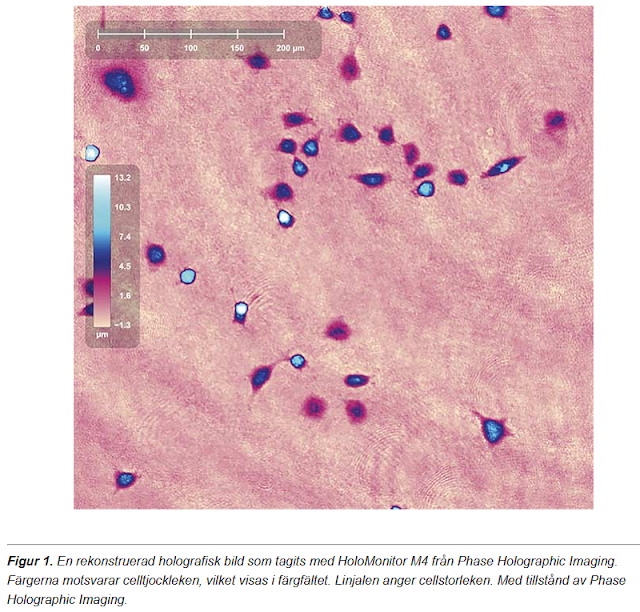



Denna teknik är på väg att bli standard.
SvaraRaderaTja,med stöd av Altiums samlade försäljningsorganisation från 12 dotterbolag samt deras egna distributionskanaler kanske det inte dröjer så länge innan HoloMonitor används på bred front och sen blir en standard.
RaderaMvh the99
Instämmer. Tror vi kommer se en skarp ökning av säljet. Spår dom att gå plus 2025 så lär säljet stegra bra nog även 2024. Vissa vill säkert invänta aktieköp men då är kursen därefter också.
RaderaTack för allt jobb du gör
SvaraRadera